Cada base de datos tiene una sintaxis diferente. Conocer esta sintaxis nos va a facilitar la consulta a la base de datos y nos va a permitir recuperar registros más pertinentes.
Por ejemplo, si consultamos MedLine a través de PubMed podremos utilizar etiquetas de campo para decirle dónde queremos que nos busque los términos. Esto también se da en la búsqueda de MedLine a través de OVID o de Embase. Pero cada base de datos se consulta de una forma diferente. Además, no todas las bases de datos ofrecen las mismas opciones de búsqueda.
En el caso de Embase tenemos la opción de utilizar operadores de proximidad (NEAR y NEXT) para indicarle a la base de datos que queremos que dos términos estén juntos o próximos entre sí:
- NEAR/n busca términos entre el número especificado de palabras (n) entre cada uno de los términos y en cualquier dirección: therapy NEAR/5 sleep busca la palabra therapy separada un máximo de 5 palabras de la palabra sleep (en cualquier orden).
- NEXT/n igual que NEAR, pero aquí es obligatorio que las palabras se encuentren en el orden indicado: no devuelve lo mismo NEAR que NEXT
En OVID también tenemos un operador de proximidad: adj:
- adj1: los dos términos juntos, en cualquier orden
- adj2: los dos términos juntos, en cualquier orden, separados por un máximo de una palabra
- adj3: los dos términos juntos, en cualquier orden, separados por un máximo de dos palabras
Hasta hoy PubMed era la única base de datos que no ofrecía operadores de proximidad, por lo que la única forma que teníamos era el uso de comillas para buscar una frase concreta: «sleep therapy» o bien buscar para que aparecieran los dos términos, independientemente del orden y del número de palabras que los separasen: sleep AND therapy. El problema de esto es la cantidad de ruido documental (registros no relevantes) que se recuperaban.
Pero esto ha cambiado: a partir de ahora PubMed permite la búsqueda de proximidad.
Esta búsqueda sólo se puede hacer en dos campos: en título y en resumen, y se especifica dentro de los operadores de campo [title] [ti] y [title/abstract] [tiab]. Su funcionamiento es similar al operador de proximidad de OVID, es decir, buscará los términos separados por el número de palabras especificadas, indistintamente del orden de las mismas.
Vamos a escribir [title:~0] cuando buscamos los dos términos seguidos, en cualquier orden:
«rationing healthcare»[title:~0] nos devolverá cosas así:

Si buscamos por «rationing healthcare»[title:~1] recuperaremos cosas así:

Para crear el operador de proximidad se utiliza el símbolo ~, que puedes crear pulsando las teclas Alt+126 en un teclado de PC o Alt+ñ en un teclado de Mac. La sintaxis es: «término término»[operadore de campo:~n] donde puedes cambiar operador de campo por [title] o la forma abreviada [ti] o por [title/abstract] o la forma abreviada [tiab] y donde la n se refiere al número de palabras que habrá entre un término y otro. Si escribimos un 0 PubMed entiende que han de ir juntas. No hay límite en el número de términos que se pueden incluir, aunque lo habitual es que se utilicen sólo dos.
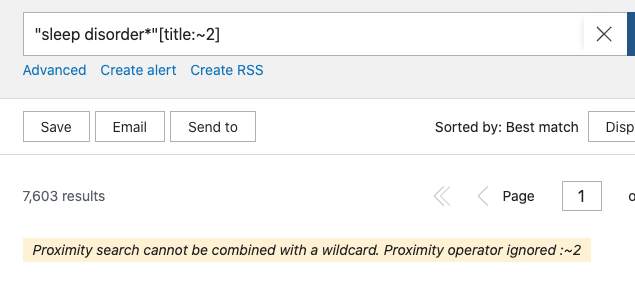
Ten en cuenta que el uso de este operador de proximidad inhibe la búsqueda automática que hace PubMed, por lo que, por ejemplo, no buscará por el descriptor MeSH o las variantes de la palabra. Tampoco permite el uso del operador de truncamiento, así que la búsqueda «sleep disorder*»[title:~2] no funcionará e ignorará el operador de proximidad.
Como siempre, si tenéis cualquier duda sobre la sintaxis de búsqueda en cualquier base de datos, y ahora especialmente sobre este tema, podéis consultar con vuestra bibliotecaria de cabecera. O contactad con Alter Biblio si no contáis con biblioteca propia.